ProfAff
Development of an Affinity Interactomic Database
Contexte scientifique
Les complexes moléculaires jouent un rôle central dans les processus cellulaires et peuvent être étudiés via des approches ciblées, permettant une compréhension approfondie d’un système moléculaire donné. Cependant, l’interactomique, qui explore l’ensemble des réseaux d’interactions moléculaires, reste aujourd’hui largement qualitative. Les réseaux sont souvent décrits sous forme de graphes, sans quantification précise des affinités entre partenaires moléculaires, limitant ainsi la compréhension fine des dynamiques cellulaires et des mécanismes biologiques.Pour surmonter cette limitation, l’équipe Quantitative Interactomics and Disease-Related Networks, fondée en 2024 à l’iBV, développe des approches novatrices de biochimie systémique permettant de cartographier simultanément à grande échelle les interactions protéiques et leurs affinités moléculaires. Ces travaux ont déjà généré un vaste ensemble de données incluant plus de 5 millions d’affinités protéiques, permettant de modéliser les différences entre réseaux d’interactions dans divers contextes cellulaires et de prédire les propriétés d’auto-organisation des réseaux. L’objectif ambitieux de l’équipe est d’atteindre 500 millions de mesures dans les années à venir.
Pour centraliser ces données, une base de données pionnière, ProfAff (Profiling Affinities), a été développée.
Objectifs du projet
Le projet Development of an Affinity Interactomic Database vise à concevoir des méthodes expérimentales et analytiques innovantes pour l’étude des interactions protéiques. En combinant les forces de la biochimie quantitative et de l’interactomique, il ambitionne de créer une base de données exhaustive référençant les interactions protéiques et leurs affinités.Les étapes clés de ce projet, en partenariat avec la MSI, incluent :
- Le développement d’un workflow informatique pour automatiser le prétraitement des données ;
- L’adaptation de la base de données ProfAff pour le stockage et l’analyse de vastes ensembles de données ;
- Le déploiement de la base de données, hébergée par Université Côte d’Azur.
Impact attendu
Le projet aboutira à une base de données publique, quantitative et exhaustive, transformant l’exploration des réseaux d’interactions protéiques. Cette ressource simplifiera l’étude des processus biologiques et pathologiques, tout en ouvrant de nouvelles perspectives pour la recherche biomédicale et le développement thérapeutique.Porteur du projet
Gergo Gogl, Responsable de l’équipe Quantitative Interactomics and Disease-Related Networks, iBV – Institut de Biologie de Valrose, Université Côte d’Azur.Participants au projet
Milissa Abboute, Stagiaire. Etudiante en Master 1, Université Côte d’Azur.Carole Belliardo, Ingénieure Opérationnelle en Calcul Intensif, Simulation Numérique et Science des Données, MSI - Maison de la Modélisation, de la Simulation et des Interactions, Université Côte d’Azur.
Boglarka Zambo, Chercheur Postdoctoral à l’iBV – Institut de Biologie de Valrose, Université Côte d’Azur.
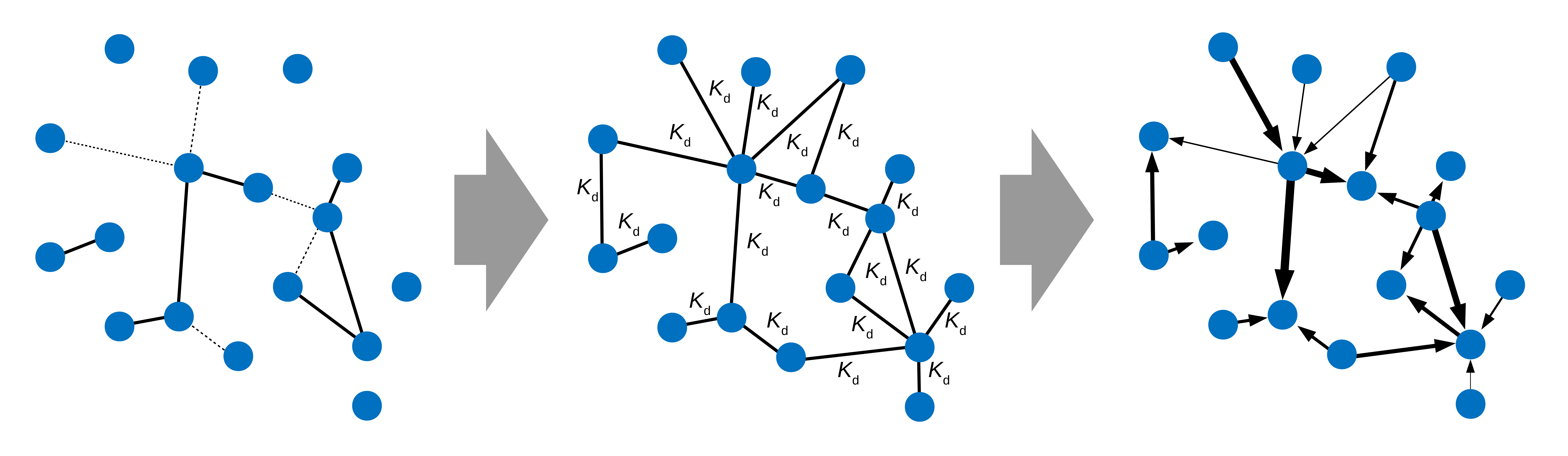
Les réseaux d'interactions sont généralement étudiés à l'aide d'outils qualitatifs qui ne permettent de capturer que les interactions les plus stables.
La biochimie systémique permet d'étudier des réseaux complets et d'explorer les paramètres de liaison qui les traversent. Ces propriétés biochimiques offrent la possibilité d'examiner des réseaux complexes sous un aspect fonctionnel.
La biochimie systémique permet d'étudier des réseaux complets et d'explorer les paramètres de liaison qui les traversent. Ces propriétés biochimiques offrent la possibilité d'examiner des réseaux complexes sous un aspect fonctionnel.

