Diagnostic des maladies mitochondriales
Développement d’une approche de médecine personnalisée pour améliorer le diagnostic des maladies mitochondriales est le sujet de recherche sur lequel Justine Labory travaille dans le cadre de son doctorat financé par le projet PIA MITOMICS et mené à l'IRCAN sous la direction du Pr Sylvie Bannwarth et avec le soutien de la MSI et notamment du Dr Silvia Bottini.
Résumé
Les maladies mitochondriales (MM) sont des pathologies rares provoquées par une déficience de la chaîne respiratoire (CR) mitochondriale, qui fournit l'énergie aux cellules par phosphorylation oxydative. Ce sont les plus fréquentes des maladies métaboliques avec une incidence estimée à 1/5000 naissances. Les MM sont extrêmement hétérogènes, tant sur le plan clinique que génétique, avec un large éventail d'âges et des symptômes très différents, ce qui rend leur diagnostic très difficile.
Les MM sont dues à des variants dans des gènes impliqués dans la biogénèse mitochondriale. Bien que les mitochondries possèdent leur propre génome, plus de 1000 gènes nucléaires sont nécessaires au fonctionnement de la CR. Par conséquent, les MM peuvent être causées par des variants pathogènes affectant soit l'ADNmt, soit les gènes nucléaires. Leur diagnostic nécessite l’identification du gène responsable.
Aujourd'hui, plus de 300 gènes nucléaires sont impliqués dans les MM et la liste ne cesse de s'allonger. Malgré le développement du séquençage à haut débit (NGS), plus d’un patient sur deux est en impasse diagnostique, le gène responsable restant inconnu.
L’arrivée du séquençage de l'exome (WES) et du séquençage du génome (WGS) a considérablement accéléré l'identification de variants sur des gènes de maladies jusqu'alors inconnus. Néanmoins, leur taux de réussite pour la détection des variants responsables se situent seulement entre 25 et 50 %. Plusieurs variants détectés par le WES restent des variants de signification inconnue (VUS) ou sont omis en raison de l'impossibilité de les classer par ordre de priorité. En outre, les variants présents dans des régions non codantes ne peuvent pas être identifiés par WES. Le WGS détecte les variants dans les régions codantes et non codantes, mais il présente un défi important (et actuellement non résolu) d'interprétation des données pour les plus de 3 millions de variants par échantillon.
Des études récentes ont montré l'intérêt du séquençage de l'ARN (RNAseq), pour sélectionner des gènes candidats présentant une expression altérée ou monoallélique et pour identifier des variants introniques profonds affectant l'épissage, lorsqu'il est utilisé en combinaison avec le WES. Pour les MM, l’utilisation du RNAseq chez 48 patients non diagnostiqués après un WES a permis d’augmenter le taux de réussite de 10%.
Dans ce contexte, le développement de nouvelles approches bio-informatiques est essentiel pour résoudre l'impasse diagnostique et améliorer notre connaissance des maladies mitochondriales.
Partenaires
- Université d’Angers
- Université Côte d’Azur
- IRCAN
- Centre Inria d'Université Côte d'Azur
- Université de Nantes
- CHU d’Angers
- CHU de Nice
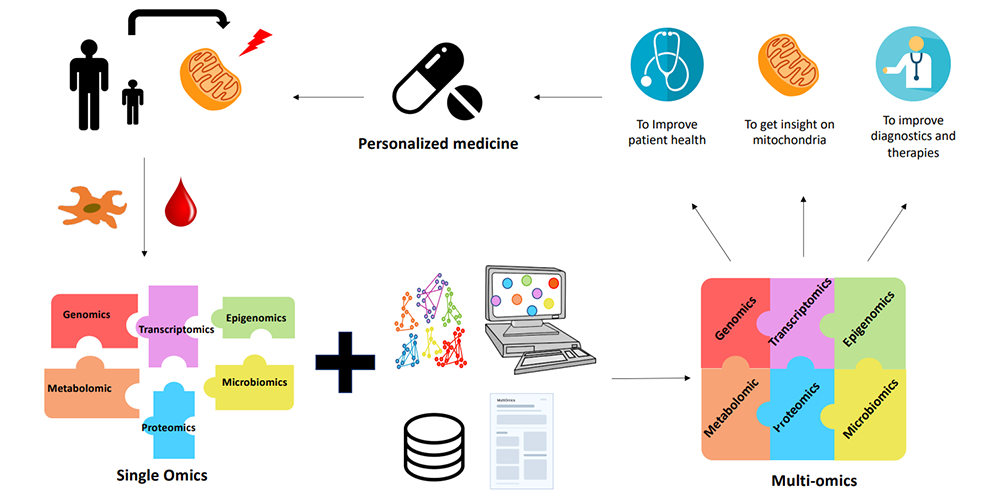
Image : L'apport de la multiomique pour les MM. Des échantillons de sang ou de tissus (biopsie de peau ou de muscle) sont prélevés sur des patients atteints de MM. Des fibroblastes ou des myotubes sont préparés et cultivés à partir de biopsies de tissus. Différentes analyses omiques sont effectuées séparément afin d'identifier le(s) gène(s) responsable(s) de la pathologie. Cependant, ces analyses ne sont pas toujours concluantes. Malgré la validité de l'approche mono-omique, la complexité des MM limite le succès du diagnostic. C'est là qu'intervient la multi-omique. Grâce à la bioinformatique et au développement d'algorithmes, l'intégration de données provenant de plusieurs analyses omiques différentes est possible et permet d'obtenir de meilleurs résultats. La multi-omique combinée aux bases de données et à la littérature permet de mieux comprendre les maladies mitochondriales. Leur diagnostic est donc amélioré et un traitement personnalisé peut être proposé pour chaque patient.

