MetaBinMG
Le projet MetaBinMG s'inscrit dans les travaux de recherche du consortium des Projets de recherche prioritaires Microbiomes de plantes cultivées et technologies numériques (PEPR MISTIC), visant à développer des outils pour étudier la diversité microbienne associée aux cultures végétales et à leur santé.
Les travaux préliminaires révèlent que les stratégies d'analyse actuelles ne parviennent pas à suffisamment représenter les génomes des communautés microbiennes des sols. En effet, les différentes technologies de séquençage disponibles semblent ne saisir qu'une fraction de la diversité réelle de ces échantillons environnementaux complexes. De plus, les méthodes bioinformatiques n'offrent qu'une reconstruction partielle des génomes microbiens.
Cette mission a un double objectif. D'une part, pour améliorer la reconstruction de génomes, il s'agit de développer des stratégies innovantes pour exploiter pleinement l'information contenue dans les diverses données métagénomiques déjà disponibles. D'autre part, il est prévu d'utiliser des données de métabarcoding en cours de production pour évaluer la part de diversité capturée par les données de type métagénomiques.
Ces résultats permettront de dimensionner précisément les efforts de séquençage métagénomique nécessaires afin de reconstruire un ensemble de génomes représentatif de la communauté de microorganismes des échantillons environnementaux.
Porteur du Projet :
Etienne Danchin, Institut Sophia Agrobiotech
Partenaires du projet :
- Institut Sophia Agrobiotech (Etienne Danchin, Marc Bailly-Bechet)
- MSI – Maison de la Modélisation, de la Simulation et des Interactions (Carole Belliardo)
- Centre Inria de l’Université de Bordeaux (David Sherman, Clémence Frioux et Alain Franc de l’équipe projet Pleiade, et Erwan Grichoux, responsable de la plateforme PGTB)
- Centre Inria de l’Université de Rennes (Nicolas Maurice, Claire Lemaitre et Riccardo Vicedomini)
- Centre INRAE Bourgogne-Franche-Comté (Samuel Mondy, responsable de la plateforme Genosol)
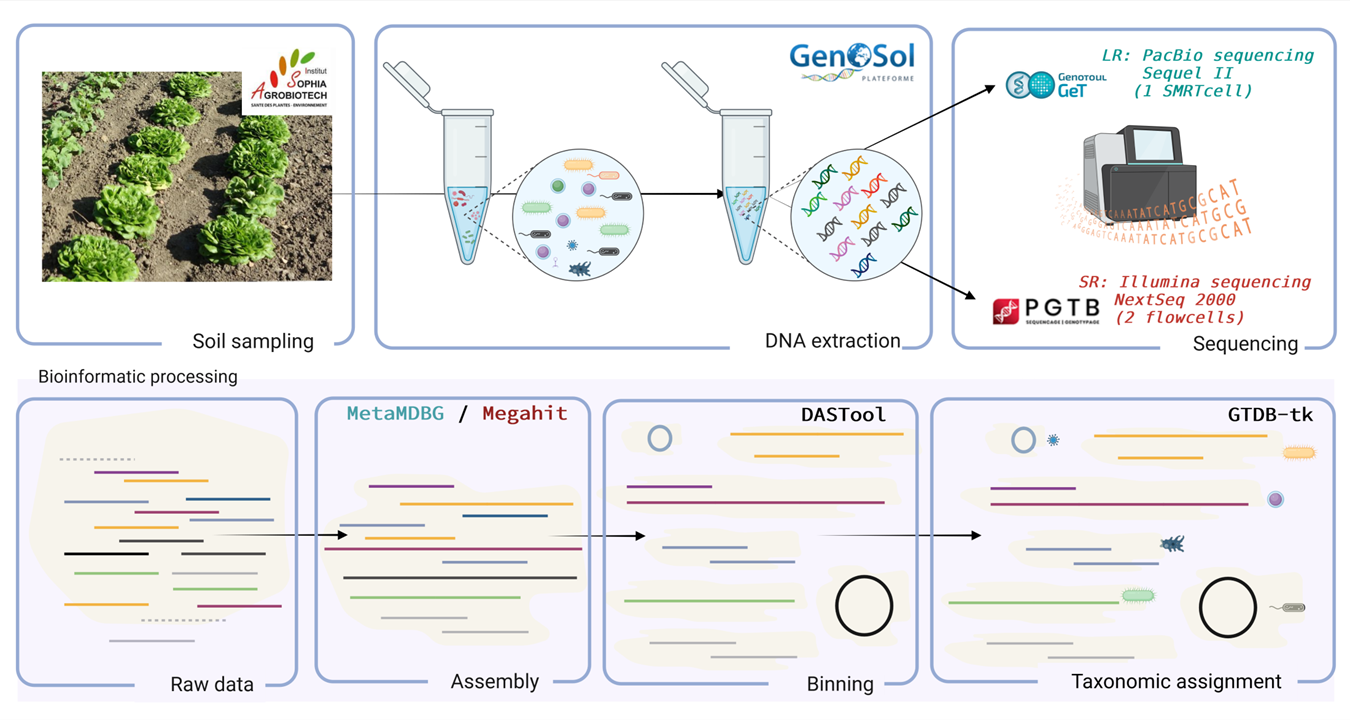
Légende
Protocole d'analyse métagénomique de sol pour la reconstruction des génomes des espèces microbiennes présentes dans un échantillon.
- Collecte d'un échantillon de sol réalisé sur une parcelle cultivée dans le sud de la France par les équipes de l'Institut Sophia Agrobiotech.
- Extraction des molécules d'ADN par la plateforme Genosol.
- Séquençage de l'ADN :
- Séquençage long-reads : PacBio HiFi Sequel II (1 flowcell) par la plateforme GetPlage de l'INRAe de Toulouse ;
- Séquençage short-reads : Illumina NextSeq 2000 2x150 pb par la plateforme PGTB de l'INRAe de Bordeaux
- Analyse bioinformatique :
- Contrôle qualité et analyse des données brutes pour éliminer les séquences erronées ou de mauvaise qualité.
- Reconstruction des génomes : Les reads de haute qualité sont ensuite assemblés pour reconstruire les génomes des micro-organismes présents dans l'échantillon.
- Regroupement (binning) des fragments d'ADN assemblés pour les rassembler par génomes. Cela permet d'associer les séquences selon l'organisme d'origine.
- Analyse taxonomique pour identifier les espèces présentes dans l'échantillon. Cette étape utilise des bases de données de référence et des méthodes de recherche d'homologie et de placement phylogénétique pour classifier les micro-organismes et comprendre la diversité microbienne du sol étudié.

